Tässä viestissä osoitamme, kuinka tehokkaasti hienosäätää huippumoderni proteiinikielimalli (pLM) ennustamaan proteiinin solunsisäistä lokalisointia käyttämällä Amazon Sage Maker.
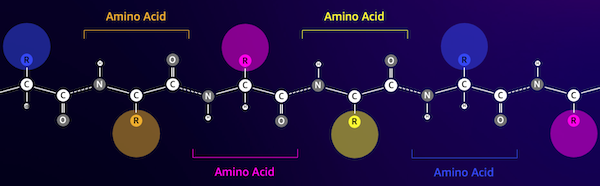
Proteiinit ovat kehon molekyylikoneita, jotka ovat vastuussa kaikesta lihasten liikuttamisesta infektioihin reagoimiseen. Tästä lajikkeesta huolimatta kaikki proteiinit koostuvat toistuvista molekyyliketjuista, joita kutsutaan aminohapoiksi. Ihmisen genomi koodaa 20 standardiaminohappoa, joista jokaisella on hieman erilainen kemiallinen rakenne. Näitä voidaan esittää aakkosten kirjaimilla, jolloin voimme analysoida ja tutkia proteiineja tekstijonona. Valtava määrä proteiinisekvenssejä ja rakenteita antaa proteiineille niiden laajan käyttötarkoituksen.
Proteiineilla on myös keskeinen rooli lääkekehityksessä mahdollisina kohteina, mutta myös terapeuttisina aineina. Kuten seuraavasta taulukosta käy ilmi, monet vuonna 2022 myydyimmistä lääkkeistä olivat joko proteiineja (etenkin vasta-aineita) tai muita molekyylejä, kuten mRNA:ta, jotka muunnettiin proteiineihin kehossa. Tämän vuoksi monien biotieteen tutkijoiden on vastattava proteiineja koskeviin kysymyksiin nopeammin, halvemmalla ja tarkemmin.
| Nimi | Valmistaja | 2022 maailmanlaajuinen myynti (miljardeja USD) | Merkintöjen |
| Yhteisöllinen | Pfizer / BioNTech | $40.8 | Covid-19 |
| Spikevax | nykyaikainen | $21.8 | Covid-19 |
| Humira | Abbvie | $21.6 | Niveltulehdus, Crohnin tauti ja muut |
| keytruda | Merck | $21.0 | Erilaisia syöpiä |
Tietolähde: Urquhart, L. Parhaat yritykset ja lääkkeet myynnin mukaan vuonna 2022. Nature Reviews Drug Discovery 22, 260–260 (2023).
Koska voimme esittää proteiineja merkkisekvensseinä, voimme analysoida niitä alun perin kirjoitetulle kielelle kehitetyillä tekniikoilla. Tämä sisältää suuret kielimallit (LLM), jotka on esiopetettu valtaville tietojoukoille, joita voidaan sitten mukauttaa tiettyihin tehtäviin, kuten tekstin yhteenvetoon tai chatboteihin. Samoin pLM:t on esikoulutettu suurissa proteiinisekvenssitietokantoissa käyttämällä leimaamatonta, itsevalvottua oppimista. Voimme mukauttaa niitä ennustamaan asioita, kuten proteiinin 3D-rakennetta tai kuinka se voi olla vuorovaikutuksessa muiden molekyylien kanssa. Tutkijat ovat jopa käyttäneet pLM:itä uusien proteiinien suunnitteluun tyhjästä. Nämä työkalut eivät korvaa ihmisten tieteellistä asiantuntemusta, mutta niillä on potentiaalia nopeuttaa esikliinistä kehitystä ja kokeiden suunnittelua.
Yksi haaste näissä malleissa on niiden koko. Sekä LLM:t että pLM:t ovat kasvaneet suuruusluokkaa viime vuosina, kuten seuraavasta kuvasta ilmenee. Tämä tarkoittaa, että niiden kouluttaminen riittävään tarkkuuteen voi kestää kauan. Se tarkoittaa myös, että malliparametrien tallentamiseen on käytettävä laitteistoa, erityisesti GPU:ita, joissa on paljon muistia.
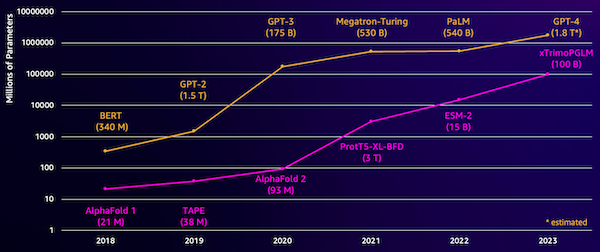
Pitkät koulutusajat ja suuret tapaukset vastaavat korkeita kustannuksia, mikä voi jättää tämän työn monien tutkijoiden ulottumattomiin. Esimerkiksi vuonna 2023 a tutkimusryhmä kuvasi 100 miljardin parametrin pLM:n kouluttamista 768 A100-grafiikkasuorittimella 164 päivän ajan! Onneksi monissa tapauksissa voimme säästää aikaa ja resursseja mukauttamalla olemassa olevaa pLM:ää erityiseen tehtäväämme. Tätä tekniikkaa kutsutaan hienosäätö, ja antaa meille myös mahdollisuuden lainata edistyneitä työkaluja muun tyyppisistä kielimallinnuksista.
Ratkaisun yleiskatsaus
Erityinen ongelma, jota käsittelemme tässä viestissä, on subsellulaarinen lokalisointi: Voimmeko proteiinisekvenssin perusteella rakentaa mallin, joka voi ennustaa, elääkö se solun ulkopuolella (solukalvo) vai sisällä? Tämä on tärkeä tieto, joka voi auttaa meitä ymmärtämään toiminnan ja olisiko siitä hyvä lääkekohde.
Aloitamme lataamalla julkisen tietojoukon käyttämällä Amazon SageMaker Studio. Sitten käytämme SageMakeria ESM-2-proteiinikielimallin hienosäätämiseen tehokkaalla harjoitusmenetelmällä. Lopuksi käytämme mallia reaaliaikaisena päättelypäätepisteenä ja käytämme sitä joidenkin tunnettujen proteiinien testaamiseen. Seuraava kaavio havainnollistaa tätä työnkulkua.
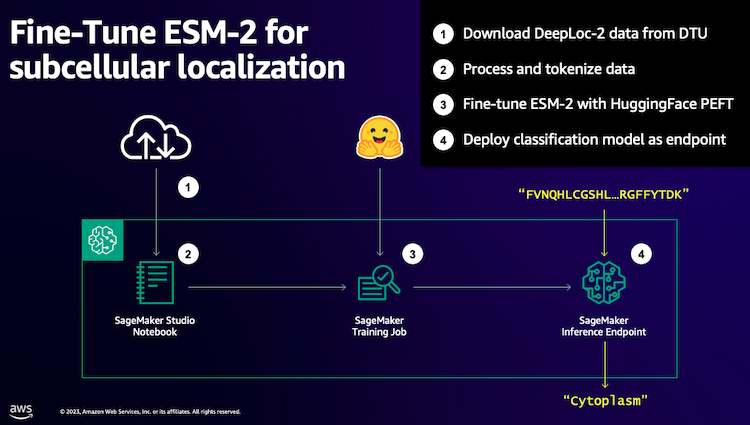
Seuraavissa osioissa käymme läpi vaiheet harjoitustietojesi valmisteluun, harjoitusskriptin luomiseen ja SageMaker-harjoitustyön suorittamiseen. Kaikki tässä viestissä oleva koodi on saatavilla osoitteessa GitHub.
Valmistele harjoitustiedot
Käytämme osaa DeepLoc-2-tietojoukko, joka sisältää useita tuhansia SwissProt-proteiineja kokeellisesti määritetyillä paikoilla. Suodatamme korkealaatuiset sekvenssit välillä 100–512 aminohappoa:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Seuraavaksi tokenisimme sekvenssit ja jaamme ne koulutus- ja arviointisarjoiksi:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Lopuksi lataamme käsitellyt koulutus- ja arviointitiedot osoitteeseen Amazonin yksinkertainen tallennuspalvelu (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Luo harjoitusskripti
SageMaker-skriptitila antaa sinun suorittaa mukautettua koulutuskoodiasi AWS:n hallinnoimissa optimoiduissa koneoppimiskehyksen (ML) säilöissä. Tätä esimerkkiä varten mukautamme an olemassa oleva skripti tekstin luokittelua varten Hugging Facesta. Näin voimme kokeilla useita tapoja parantaa koulutustyömme tehokkuutta.
Tapa 1: Painotettu harjoitusluokka
Kuten monet biologiset tietojoukot, DeepLoc-data on jakautunut epätasaisesti, mikä tarkoittaa, että kalvo- ja ei-membraaniproteiineja ei ole yhtä montaa. Voisimme ottaa uudelleen näytteitä tiedoistamme ja hylätä enemmistöluokan tietueet. Tämä kuitenkin vähentäisi harjoitustietojen kokonaismäärää ja mahdollisesti vahingoittaisi tarkkuuttamme. Sen sijaan laskemme harjoitustyön aikana luokkapainot ja käytämme niitä tappion säätämiseen.
Koulutuskäsikirjoituksessamme alaluokitamme Trainer luokasta alkaen transformers kanssa WeightedTrainer luokka, joka ottaa luokkapainot huomioon laskettaessa ristientropiahäviötä. Tämä auttaa estämään harhaa mallissamme:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMenetelmä 2: Gradientin kerääntyminen
Gradienttikertymä on koulutustekniikka, jonka avulla mallit voivat simuloida harjoittelua suuremmilla eräkokoilla. Tyypillisesti erän kokoa (yhdessä harjoitusvaiheessa gradientin laskemiseen käytettyjen näytteiden lukumäärää) rajoittaa GPU:n muistikapasiteetti. Gradientin keräämisessä malli laskee ensin gradientit pienemmille erille. Sen sijaan, että mallin painot päivitettäisiin heti, gradientit kerääntyvät useille pienille erille. Kun kertyneet gradientit ovat yhtä suuret kuin tavoitesuurempi eräkoko, optimointivaihe suoritetaan mallin päivittämiseksi. Tämän ansiosta mallit voivat harjoitella tehokkaasti isommilla erillä ylittämättä GPU-muistirajaa.
Lisälaskentaa tarvitaan kuitenkin pienemmän erän eteen- ja taaksepäin siirtymistä varten. Suuremmat eräkoot gradientin kertymisen kautta voivat hidastaa harjoittelua, varsinkin jos käytetään liian monta kertymisvaihetta. Tavoitteena on maksimoida grafiikkasuorittimen käyttö, mutta välttää liiallinen hidastuminen liian monista ylimääräisistä gradientin laskentavaiheista.
Tapa 3: Gradientin tarkistuspiste
Gradienttitarkistus on tekniikka, joka vähentää harjoituksen aikana tarvittavaa muistia ja pitää laskennallisen ajan kohtuullisena. Suuret hermoverkot vievät paljon muistia, koska niiden on tallennettava kaikki väliarvot eteenpäin kulkemisesta voidakseen laskea gradientit taaksepäin siirtymisen aikana. Tämä voi aiheuttaa muistiongelmia. Yksi ratkaisu on, että näitä väliarvoja ei tallenneta, vaan ne on laskettava uudelleen taaksepäin ajon aikana, mikä vie paljon aikaa.
Gradienttitarkistus tarjoaa tasapainoisen lähestymistavan. Se tallentaa vain osan väliarvoista, ns tarkistuspisteitä, ja laskee muut tarpeen mukaan uudelleen. Siksi se käyttää vähemmän muistia kuin kaiken tallentaminen, mutta myös vähemmän laskentaa kuin kaiken uudelleenlaskeminen. Valitsemalla strategisesti, mitkä aktivaatiot tarkistuspisteeseen, gradienttitarkistuspiste mahdollistaa suurten hermoverkkojen koulutuksen hallittavalla muistinkäytöllä ja laskenta-ajalla. Tämän tärkeän tekniikan ansiosta on mahdollista kouluttaa erittäin suuria malleja, jotka muuten joutuisivat muistirajoituksiin.
Harjoitusskriptissämme otamme gradientin aktivoinnin ja tarkistuspisteen käyttöön lisäämällä tarvittavat parametrit TrainingArguments esine:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Menetelmä 4: LLM:ien matala-arvoinen mukauttaminen
Suuret kielimallit, kuten ESM-2, voivat sisältää miljardeja parametreja, joiden kouluttaminen ja käyttäminen on kallista. Tutkijat kehitti koulutusmenetelmän nimeltä Low-Rank Adaptation (LoRA), jotta näiden valtavien mallien hienosäätö olisi tehokkaampaa.
LoRA:n perusideana on, että kun hienosäädät mallia tiettyä tehtävää varten, sinun ei tarvitse päivittää kaikkia alkuperäisiä parametreja. Sen sijaan LoRA lisää malliin uusia pienempiä matriiseja, jotka muuttavat tulot ja lähdöt. Vain nämä pienemmät matriisit päivitetään hienosäädön aikana, mikä on paljon nopeampaa ja kuluttaa vähemmän muistia. Alkuperäisen mallin parametrit pysyvät jäädytettyinä.
Hienosäädön jälkeen LoRA:lla voit yhdistää pienet mukautetut matriisit takaisin alkuperäiseen malliin. Tai voit pitää ne erillään, jos haluat nopeasti hienosäätää mallin muita tehtäviä varten unohtamatta aiempia tehtäviä. Kaiken kaikkiaan LoRA mahdollistaa LLM:ien tehokkaan mukauttamisen uusiin tehtäviin murto-osalla tavanomaisista kustannuksista.
Harjoitusskriptissämme määritämme LoRA:n käyttämällä PEFT Hugging Facen kirjasto:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Lähetä SageMaker-koulutustyö
Kun olet määrittänyt koulutusohjelman, voit määrittää ja lähettää SageMaker-harjoitustyön. Määritä ensin hyperparametrit:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Määritä seuraavaksi, mitä mittareita harjoituslokeista kerätään:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Määrittele lopuksi Hugging Face -estimaattori ja lähetä se koulutukseen ml.g5.2xlarge ilmentymätyypillä. Tämä on kustannustehokas ilmentymätyyppi, joka on laajalti saatavilla monilla AWS-alueilla:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)Seuraavassa taulukossa verrataan eri koulutusmenetelmiä, joista keskustelimme, ja niiden vaikutusta työmme suoritusaikaan, tarkkuuteen ja GPU-muistivaatimuksiin.
| Konfigurointi | Laskutettava aika (min) | Arvioinnin tarkkuus | Suurin GPU-muistin käyttö (GB) |
| Perusmalli | 28 | 0.91 | 22.6 |
| Base + GA | 21 | 0.90 | 17.8 |
| Pohja + GC | 29 | 0.91 | 10.2 |
| Pohja + LoRA | 23 | 0.90 | 18.6 |
Kaikki menetelmät tuottivat malleja korkealla arviointitarkkuudella. LoRA:n ja gradienttiaktivoinnin käyttö vähensi suoritusaikaa (ja kustannuksia) 18 % ja 25 %. Gradienttitarkistuspisteiden käyttö vähensi grafiikkasuorittimen enimmäismuistin käyttöä 55 %. Rajoituksistasi (kustannus, aika, laitteisto) riippuen yksi näistä lähestymistavoista voi olla järkevämpi kuin toinen.
Jokainen näistä menetelmistä toimii hyvin yksinään, mutta mitä tapahtuu, kun käytämme niitä yhdessä? Seuraavassa taulukossa on yhteenveto tuloksista.
| Konfigurointi | Laskutettava aika (min) | Arvioinnin tarkkuus | Suurin GPU-muistin käyttö (GB) |
| Kaikki menetelmät | 12 | 0.80 | 3.3 |
Tässä tapauksessa näemme tarkkuuden heikkenemisen 12 %. Olemme kuitenkin vähentäneet suoritusaikaa 57 % ja GPU-muistin käyttöä 85 %! Tämä on valtava lasku, jonka ansiosta voimme harjoitella monenlaisia kustannustehokkaita ilmentymätyyppejä.
Puhdistaa
Jos seuraat asiaa omalla AWS-tililläsi, poista kaikki reaaliaikaiset päättelypäätepisteet ja luomasi tiedot välttääksesi lisäveloitukset.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Yhteenveto
Tässä viestissä osoitimme, kuinka tehokkaasti hienosäätää proteiinikielimalleja, kuten ESM-2, tieteellisesti merkityksellistä tehtävää varten. Lisätietoja Transformers- ja PEFT-kirjastojen käyttämisestä pLMS:n kouluttamiseen on viesteissä Syväoppiminen proteiinien kanssa ja ESMBind (ESMB): ESM-2:n matalan tason mukautus proteiinia sitovan kohdan ennustamiseen Hugging Face -blogissa. Löydät myös lisää esimerkkejä koneoppimisen käyttämisestä proteiinien ominaisuuksien ennustamiseen Mahtava AWS:n proteiinianalyysi GitHub-arkisto.
kirjailijasta
 Brian uskollinen on vanhempi AI/ML-ratkaisuarkkitehti maailmanlaajuisessa terveydenhuollon ja biotieteiden tiimissä Amazon Web Services -palvelussa. Hänellä on yli 17 vuoden kokemus biotekniikasta ja koneoppimisesta, ja hän on intohimoinen auttamaan asiakkaita ratkaisemaan genomi- ja proteomihaasteita. Vapaa-ajallaan hän nauttii ruoanlaitosta ja syömisestä ystäviensä ja perheensä kanssa.
Brian uskollinen on vanhempi AI/ML-ratkaisuarkkitehti maailmanlaajuisessa terveydenhuollon ja biotieteiden tiimissä Amazon Web Services -palvelussa. Hänellä on yli 17 vuoden kokemus biotekniikasta ja koneoppimisesta, ja hän on intohimoinen auttamaan asiakkaita ratkaisemaan genomi- ja proteomihaasteita. Vapaa-ajallaan hän nauttii ruoanlaitosta ja syömisestä ystäviensä ja perheensä kanssa.
- SEO-pohjainen sisällön ja PR-jakelu. Vahvista jo tänään.
- PlatoData.Network Vertical Generatiivinen Ai. Vahvista itseäsi. Pääsy tästä.
- PlatoAiStream. Web3 Intelligence. Tietoa laajennettu. Pääsy tästä.
- PlatoESG. hiili, CleanTech, energia, ympäristö, Aurinko, Jätehuolto. Pääsy tästä.
- PlatonHealth. Biotekniikan ja kliinisten kokeiden älykkyys. Pääsy tästä.
- Lähde: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/